TIANSeq адаптер с един индекс (Illumina)
Характеристика
■ Удобно: SetA и SetB предлагат 24 единични индекса, които могат да се избират свободно според изискванията.
■ Лесен за използване: Комплектът е оборудван с буфер за разреждане на адаптера, който има силна стабилност и може да се използва директно за разреждане на адаптерен разтвор.
■ Контрол на качеството: Строгият контрол на качеството и функционалната проверка между партидите гарантират точността на индексната последователност.
Приложения
1. Този продукт се използва за изграждане на ДНК и РНК библиотеки за високопроизводителна платформа за секвениране на Illumina при прилагане на секвениране от следващо поколение (NGS).
2. Специфичните приложения на продукта включват секвениране на цял екзон, насочено секвениране, RNA-Seq, ChIP-Seq, насочено секвениране и секвениране на целия геном.
3. Трябва да се отбележи, че този продукт не е подходящ за секвениране, свързано с метилиране.
Информация за последователността на адаптера
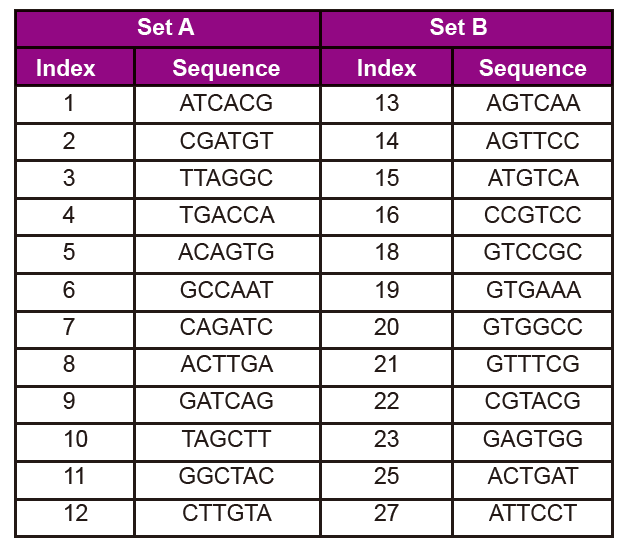
Последователността на адаптера включва следната информация:
1. Универсална последователност
5'-AATGATACGGCGACCACCGAGATCTACACTCTTTCCCT
ACACGACGCTCTTCCGATCT-3 '
2. Индекс, включващ последователност
5'-GATCGGAAGAGCACACGTCTGAACTCCAGTCAC [индекс1-27] ATCTCGTATGCCGTCTTCTGCTTG-3 '
3. Номер и последователност на индекса
Всички продукти могат да бъдат персонализирани за ODM/OEM. За детайли,моля, щракнете върху Персонализирана услуга (ODM/OEM)
Понастоящем технологията за секвениране с висока производителност се основава главно на технологията за секвениране от следващо поколение. Тъй като дължината на четене на технологията за секвениране от следващо поколение е ограничена, трябва да разделим последователността на пълната дължина на малки библиотеки с фрагменти до последователност. Според нуждите на различни експерименти за секвениране обикновено избираме секвениране с единичен край или секвениране с двоен край. Понастоящем ДНК фрагментите от библиотеката за секвениране от следващо поколение обикновено са разпределени в диапазона от 200-800 bp.

а) ДНК е с лошо качество и съдържа инхибитори. Използвайте висококачествени ДНК проби, за да избегнете инхибиране на ензимната активност.
б) Количеството ДНК проба е недостатъчно, когато се използва метод без PCR за изграждане на ДНК библиотека. Когато въвеждането на фрагментираната ДНК надвишава 50 ng, работният процес без PCR може да бъде селективно проведен по време на процеса на изграждане на библиотеката. Ако номерът на копието на библиотеката е твърде нисък, за да бъде директно секвениран, ДНК библиотеката може да бъде амплифицирана чрез PCR след лигирането на адаптера.
в) Замърсяването с РНК води до неточно първоначално количествено определяне на ДНК Замърсяването с РНК може да съществува в процеса на пречистване на геномна ДНК, което може да доведе до неточно количествено определяне на ДНК и недостатъчно зареждане на ДНК по време на изграждането на библиотеката. РНК може да се отстрани чрез третиране с РНКаза.
А-1
а) Появяват се малки фрагменти (60 bp-120 bp). Малки фрагменти обикновено са фрагменти от адаптер или димери, образувани от адаптери. Пречистването с магнитни мъниста Agencourt AMPure XP може ефективно да премахне тези фрагменти от адаптера и да гарантира качеството на последователността.
б) Големи фрагменти се появяват в библиотеката след PCR амплификация. Размерът на библиотечния ДНК фрагмент ще се увеличи със 120 bp след лигирането на адаптера. Ако фрагментът на ДНК се увеличи с повече от 120 bp след лигирането на адаптера, това може да бъде причинено от анормална амплификация на фрагмента от прекомерна PCR амплификация. Намаляването на броя на PCR циклите може да предотврати ситуацията.
в) Ненормален размер на библиотечните ДНК фрагменти след свързване на адаптера Дължината на адаптера в този комплект е 60 bp. Когато двата края на фрагмента се лигират към адаптерите, дължината ще се увеличи само със 120 bp. Когато използвате адаптер, различен от този, предоставен в този комплект, моля, свържете се с доставчика, за да предоставите съответна информация, като например дължината на адаптера. Моля, уверете се, че работният процес и операцията на експеримента следват стъпките, описани в ръководството.
г) Ненормален размер на ДНК фрагмента преди лигирането на адаптера Причината за този проблем може да бъде причинена от неправилни условия на реакция по време на фрагментацията на ДНК. За различно въвеждане на ДНК трябва да се използват различни времена на реакция. Ако входящата ДНК е повече от 10 ng, препоръчваме да изберете времето за реакция от 12 минути като начално време за оптимизация, а размерът на фрагмента, произведен по това време, е главно в диапазона от 300-500 bp. Потребителите могат да увеличат или намалят дължината на фрагментите на ДНК за 2-4 минути според собствените си изисквания, за да оптимизират фрагментите на ДНК с необходимия размер.
А-2
а) Времето за фрагментиране не е оптимизирано Ако фрагментираната ДНК е твърде малка или твърде голяма, моля, вижте Насоките за избор на време за фрагментиране, предоставени в инструкцията за определяне на времето за реакция, и използвайте тази времева точка като контрол, допълнително настройте реакционната система за удължаване или съкращаване на 3 минути, за да се направи по -точна настройка на времето за фрагментация.
А-3
Ненормално разпределение на размера на ДНК след третиране с фрагментация
а) Неправилен метод на размразяване на реагента за фрагментиране или реагентът не е напълно смесен след размразяване. Размразете 5 × реагента за раздробяване на ензимната смес върху лед. След размразяване разбъркайте реагента равномерно, като внимателно прелиствате дъното на епруветката. Не вихрете реактива!
б) Входната проба на ДНК съдържа EDTA или други замърсители. Изчерпването на солените йони и хелатиращите агенти в етапа на пречистване на ДНК е особено важно за успеха на експеримента. Ако ДНК се разтвори в 1 × TE, използвайте метода, предоставен в инструкцията, за извършване на фрагментация. Ако концентрацията на EDTA в разтвора е несигурна, се препоръчва да се пречисти ДНК и да се разтвори в дейонизирана вода за последваща реакция.
в) Неточно първоначално количествено определяне на ДНК Размерът на фрагментираната ДНК е тясно свързан с количеството на вложената ДНК. Преди третирането на фрагментиране, точното количествено определяне на ДНК с помощта на Qubit, Picogreen и други методи е от съществено значение за определяне на точното количество ДНК в реакционната система.
г) Подготовката на реакционната система не следва инструкциите Подготовката на фрагментирана реакционна система трябва да се извършва върху лед стриктно съгласно инструкциите. За да се осигури най -добър ефект, всички компоненти на реакцията трябва да бъдат поставени върху лед и подготовката на реакционната система трябва да се извърши след пълно охлаждане. След като подготовката приключи, моля, натиснете или пипетирайте, за да разбъркате добре. Не вихрете!
1. Неправилният метод на смесване (вихър, силни трептения и т.н.) ще доведе до ненормално разпределение на библиотечните фрагменти (както е показано на следващата фигура), като по този начин ще се отрази на качеството на библиотеката. Ето защо, когато приготвяте реакционния разтвор на Fragmentation Mix, моля, пипетирайте внимателно нагоре и надолу, за да смесите, или използвайте върха на пръста, за да разбъркате и разбъркате равномерно. Внимавайте да не се смесвате с вихър.

2. ДНК с висока чистота трябва да се използва за изграждане на библиотека
■ Добра целостта на ДНК: Електрофорезната лента е повече от 30 kb, без опашки
■ OD260/230:> 1.5
■ OD260/280: 1,7-1,9
3. Количеството на въведената ДНК трябва да е точно Препоръчва се използването на методите Qubit и PicoGreen за количествено определяне на ДНК, вместо на Nanodrop.
4. Трябва да се определи съдържанието на EDTA в разтвор на ДНК. EDTA има голямо влияние върху реакцията на фрагментация. Ако съдържанието на EDTA е високо, трябва да се извърши пречистване на ДНК преди последващия тест.
5. Реакционният разтвор на фрагментация трябва да се приготви върху лед Процесът на фрагментация е чувствителен към реакционната температура и време (особено след добавяне на подобрител). За да се гарантира точността на времето за реакция, моля, подгответе реакционната система върху лед.
6. Времето за реакция на фрагментация трябва да бъде точно Времето за реакция на етапа на фрагментиране ще повлияе директно върху размера на фрагментните продукти, като по този начин ще повлияе на разпределението по размер на ДНК фрагментите в библиотеката.
1. Какъв тип проба е приложим за този комплект?
Приложимият тип проба от този комплект може да бъде обща РНК или пречистена иРНК с добра целостта на РНК. Ако за изграждане на библиотеката се използва обща РНК, се препоръчва първо да се използва комплект за изчерпване на рРНК (Cat#4992363/4992364/4992391), за да се премахне първо рРНК.
2. Могат ли пробите от FFPE да се използват за конструиране на библиотека с този комплект?
ИРНК в проби от FFPE ще се разгради до известна степен, с относително лоша цялост. Когато използвате този комплект за изграждане на библиотека, се препоръчва да се оптимизира времето за фрагментиране (да се съкрати времето за фрагментация или да не се извърши фрагментация).
3. Използвайки стъпката за избор на размер, предоставена в ръководството за продукта, какво може да причини вмъкнатия сегмент да изглежда леко отклонение?
Изборът на размер трябва да се извършва в строго съответствие със стъпката за избор на размер в това ръководство за продукта. Ако има отклонение, причината може да е, че магнитните перли не са балансирани до стайна температура или не са напълно смесени, пипетата не е точна или течността е останала в върха. Препоръчва се да използвате съветите с ниска адсорбция за експеримента.
4. Избор на адаптери в библиотечното строителство
Комплектът за изграждане на библиотеката не съдържа адаптер реагент и се препоръчва да се използва заедно с TIANSeq адаптер с един индекс (Illumina) (4992641/4992642/4992378).
5. QC на библиотеката
Количествено откриване на библиотеката: Qubit и qPCR се използват за определяне на масовата концентрация и моларната концентрация на библиотеката съответно. Операцията е строго в съответствие с ръководството за продукта. Концентрацията на библиотеката като цяло ще отговаря на изискванията за NGS секвениране. Откриване на обхват на разпространение на библиотеки: Използвайки Agilent 2100 Bioanalyzer за откриване на диапазона на разпространение на библиотеката.
6. Избор на номера на цикъла на усилване
Съгласно инструкциите, броят на PCR циклите е 6-12 и броят на необходимите PCR цикли трябва да бъде избран според входа на пробата. В библиотеките с висок добив свръхусилването обикновено се проявява в различна степен, което се проявява с малко по-голям пик след пика на целевия диапазон при откриването на биоанализатор Agilent 2100 или откритата концентрация на Qubit е по-ниска от тази на qPCR. Лекото усилване е нормално явление, което не се отразява на библиотечното секвениране и последващия анализ на данните.
7. В профила на откриване на биоанализатора Agilent 2100 се появяват шипове
Появата на шипове при откриване на биоанализатор Agilent 2100 се дължи на неравномерното фрагментиране на пробите, където ще има повече фрагменти с определен размер и това ще стане по -очевидно след обогатяване с PCR. В този случай се предлага да не се извършва подбор на размера, т.е. да се зададе условието за фрагментация на 94 ° C за 15 минути инкубиране, където разпределението на фрагментите е малко и концентрирано и хомогенността може да бъде подобрена.
Категории продукти
ЗАЩО ИЗБЕРЕТЕ НАС
От създаването си нашата фабрика разработва първокласни продукти с придържане към принципа
първо качество. Нашите продукти са спечелили отлична репутация в индустрията и ценно доверие сред нови и стари клиенти.








